Experimente mit mehreren Nervenzellen Teil 1¶
Umgebung vorbereiten¶
Bevor wir mit unseren Experimenten beginnen können, müssen wir erneut unsere Umgebung vorbereiten:
from _static.common.helpers import setup_hardware_client
setup_hardware_client()
Die Feuerkette¶
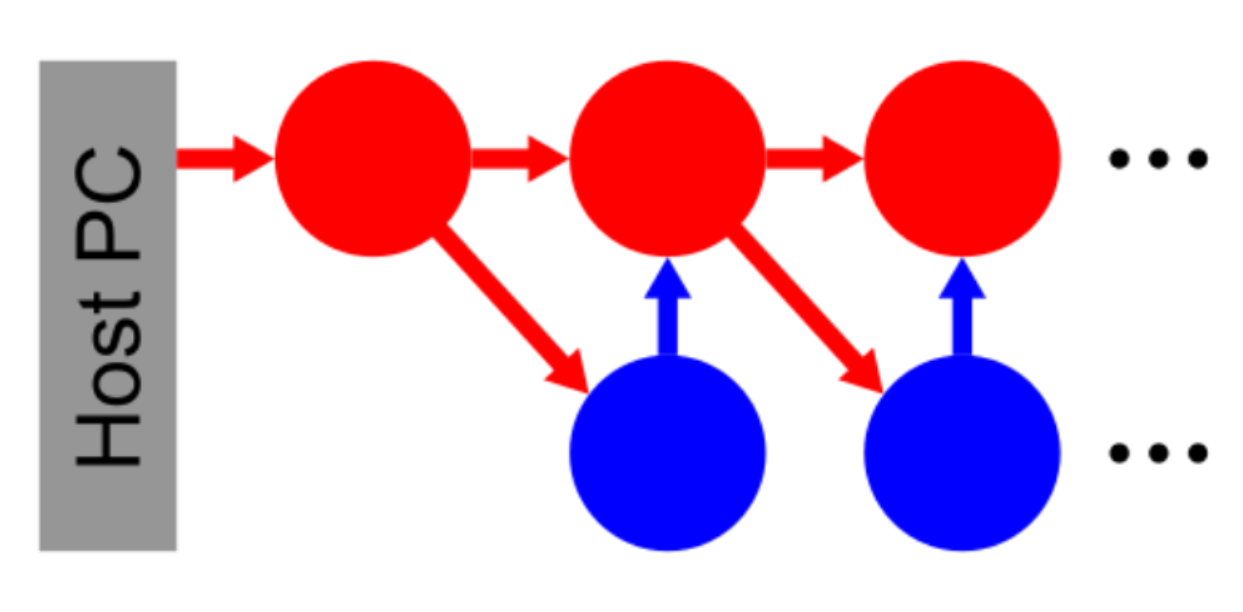
Nun wollen wir beobachten, wie sich mehrere Neuronen gemeinsam verhalten. Dazu gruppieren wir sie zu Populationen und verbinden diese zu einer Kette. Jede exzitatorische Population (rot) besitzt dabei eine inhibitorische Partner Population (blau). Auf diese Weise soll ein Signal durch eine möglichst lange Kette gereicht werden.
import pynn_brainscales.brainscales2 as pynn
%matplotlib inline
import matplotlib.pyplot as plt
import numpy as np
import ipywidgets as w
from IPython.display import HTML
from functools import partial
from _static.common.helpers import get_nightly_calibration
Slider = partial(w.IntSlider, continuous_update=False)
display(HTML('''
<style>
.jupyter-widgets { min-width: 40em !important; }
.widget-label { min-width: 30ex !important; }
</style>'''))
@w.interact(
inh_exc=Slider(30, 0, 63, description='Synaptisches Gewicht "inh_exc"'),
exc_exc=Slider(60, 0, 63, description='Synaptisches Gewicht "exc_exc"'),
exc_inh=Slider(40, 0, 63, description='Synaptisches Gewicht "exc_inh"'),
)
def experiment(inh_exc, exc_exc, exc_inh):
# Diese Parameter sind im Prinzip auch verstellbar, haben so aber gute Werte.
# Alle Gewichte können Werte im Bereich 0-63 annehmen.
weights = {"stim_exc": 60,
"stim_inh": 15}
numb_pops = 25
pop_sizes = {"exc": 5,
"inh": 5}
runtime = 0.3
closed = True
# Die Refraktärzeit muss auf einen kleinen Wert gestellt werden, um regelmäßiges
# Feuern prinzipiell zu erlauben.
neuron_parameters = {"refractory_period_refractory_time": 50}
# Die Umgebung wird vorbereitet.
calib = get_nightly_calibration()
pynn.setup(initial_config=calib)
# Die angelegten Populationen werden in einem Verzeichnis gespeichert,
# das zu Beginn leere Listen enthält.
pop_collector = {"exc": [],
"inh": []}
# Hier werden die Populationen erstellt.
for synapse_type in ["exc", "inh"]:
pop_size = pop_sizes[synapse_type]
pop = pynn.Population(pop_size * numb_pops,
pynn.cells.HXNeuron(**neuron_parameters))
pop.record(['spikes'])
for index in range(numb_pops):
mask = slice(index * pop_size, index * pop_size + pop_size)
popview = pynn.PopulationView(pop, mask)
pop_collector[synapse_type].append(popview)
# Zuerst wird ein Stimulus zum Zeitpunkt t=0 an die jeweils erste Population gegeben.
stim_pop = pynn.Population(pop_sizes['exc'],
pynn.cells.SpikeSourceArray(spike_times=[0]))
pynn.Projection(stim_pop, pop_collector['exc'][0],
pynn.AllToAllConnector(),
synapse_type=pynn.synapses.StaticSynapse(weight=weights["stim_exc"]),
receptor_type='excitatory')
pynn.Projection(stim_pop, pop_collector['inh'][0],
pynn.AllToAllConnector(),
synapse_type=pynn.synapses.StaticSynapse(weight=weights["stim_inh"]),
receptor_type='excitatory')
# Nun werden alle exzitatorischen Populationen in einer Kette miteinander verbunden.
# Außerdem besitzt jede exzitatorische Population eine inhibitorische Partnerpopulation.
# Wenn die Kette offen ist, müssen die exzitatorischen Verbindungen einmal weniger
# iteriert werden.
lastiter = numb_pops
if not closed:
lastiter -= 1
pynn.Projection(pop_collector['inh'][lastiter],
pop_collector['exc'][lastiter],
pynn.AllToAllConnector(),
synapse_type=pynn.synapses.StaticSynapse(weight=-inh_exc),
receptor_type='inhibitory')
for pop_index in range(lastiter):
pynn.Projection(pop_collector['exc'][pop_index],
pop_collector['exc'][(pop_index + 1) % numb_pops],
pynn.AllToAllConnector(),
synapse_type=pynn.synapses.StaticSynapse(weight=exc_exc),
receptor_type='excitatory')
pynn.Projection(pop_collector['exc'][pop_index],
pop_collector['inh'][(pop_index + 1) % numb_pops],
pynn.AllToAllConnector(),
synapse_type=pynn.synapses.StaticSynapse(weight=exc_inh),
receptor_type='excitatory')
pynn.Projection(pop_collector['inh'][pop_index],
pop_collector['exc'][pop_index],
pynn.AllToAllConnector(),
synapse_type=pynn.synapses.StaticSynapse(weight=-inh_exc),
receptor_type='inhibitory')
# Jetzt wird das Netzwerk emuliert.
pynn.run(runtime)
# Die Spikes werden ausgelesen.
spike_collector = {'exc': np.zeros(numb_pops, dtype=object),
'inh': np.zeros(numb_pops, dtype=object)}
for syn_type in ['exc', 'inh']:
for pop_index in range(numb_pops):
spike_collector[syn_type][pop_index] =\
pop_collector[syn_type][pop_index].get_data("spikes").segments[0].spiketrains
# Das Ergebnis wird visualisiert.
ax = plt.subplot(211)
for syn_type in spike_collector:
for pop_index in range(numb_pops):
for spike_train in spike_collector[syn_type][pop_index]:
neuron_id = spike_train.annotations['source_id']
ax.plot(spike_train, np.ones_like(spike_train) * neuron_id,
c='k', marker="o", ls="None", ms=1)
ax.set_xlim(0, runtime)
ax.set_xlabel("Zeit [ms]")
ax.set_ylim(-0.5, (pop_sizes['exc'] + pop_sizes['inh']) * numb_pops - 0.5)
ax.set_ylabel('Neuron ID')
ax.axhspan(-0.5, pop_sizes['exc'] * numb_pops - 0.5, color='r', alpha=0.2)
ax.axhspan(pop_sizes['exc'] * numb_pops - 0.5, (pop_sizes['exc'] +
pop_sizes['inh']) * numb_pops - 0.5, color='b', alpha=0.2)
ax.figure.set_size_inches(12, 15)
pynn.end()
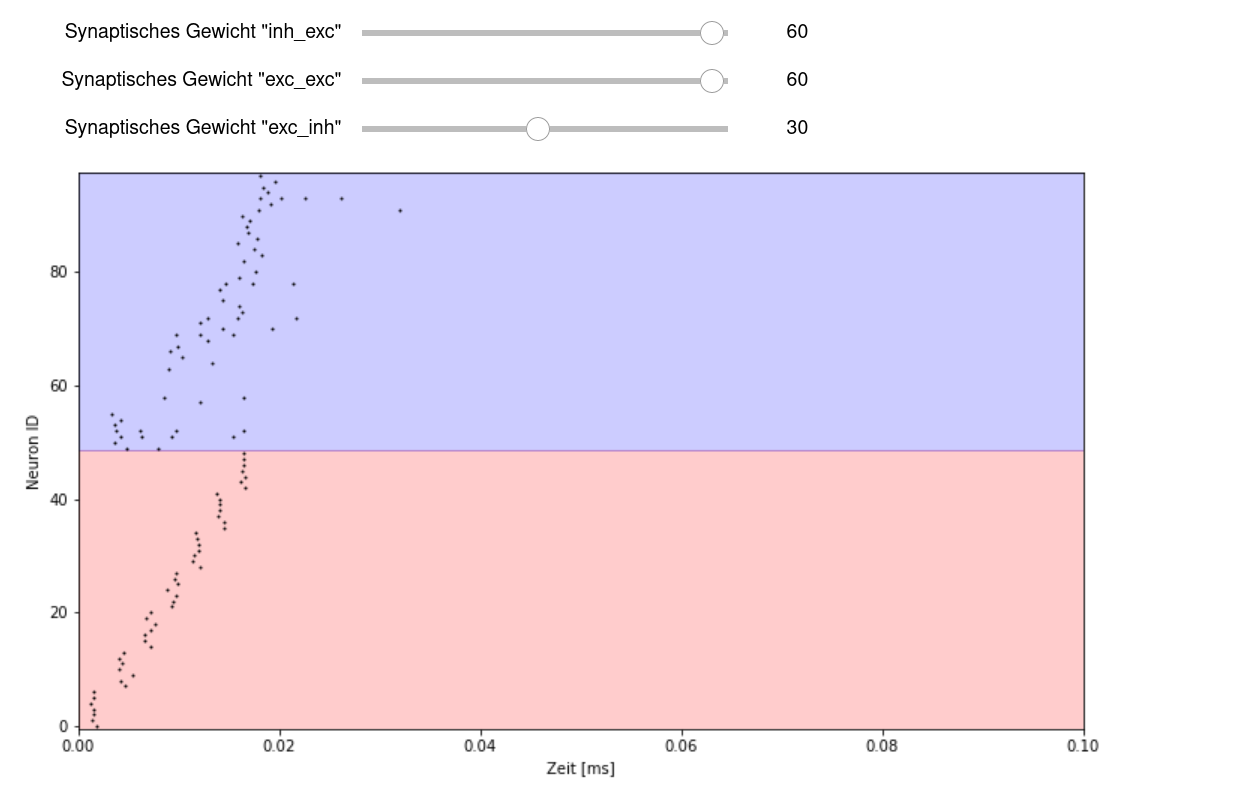
Jeder aufgetretene Spike ist durch einen Punkt gekennzeichnet. Unten im roten Bereich sind die Spikes der exzitatorischen Populationen aufgetragen, oben im blauen Bereich die der inhibitorischen Populationen. Diese sind dafür da, um das Feuern der exzitatorischen Populationen zu begrenzen.
Möglicherweise seht ihr zu Beginn ein eher aufgefächertes Bild. Das entsteht, wenn die Aktivität der Neuronen nicht stark genug unterdrückt wird. Um dies zu erreichen, kann entweder das Gewicht der inhibitorischen Synapsen
"inh_exc"erhöht werden.Wenn die exzitatorischen Populationen mehrfach feuern, bekommen sie zu viel Anregungen. Dann könnt ihr das Gewicht
"exc_exc"niedriger machen, was das weiterzugebene Signal abschwächt. Allerdings darf es nicht zu klein werden, da sonst die Kette abbricht.Es kann auch vorkommen, dass nur die Spikes der inhibitorischen Populationen verschwinden. Dann muss das Gewicht
"exc_inh"erhöht werden. Auch hier kommt es auf die Feineinstellung an: Werden die inhibitorischen Synapsen zu stark gewählt, unterdrücken sie das Signal zu sehr und die Kette bricht wieder ab.
Durch ein gutes Zusammenspiel dieser drei Gewichte ist es möglich, eine recht saubere Treppenform im roten Bereich bei den exzitatorischen Neuronen zu bekommen. Was bedeutet die Anzahl der Stufen, was ihre Höhe? Fällt euch eine Erklärung ein, warum die inhibitorischen Spikes verteilter sind?
Falls ihr die Anzahl der Populationen oder ihre Größen verändert, könnte ein Fehler auftreten. Dies ist der Fall, wenn das Limit der zur Ansteuerung verfügbaren Neuronen und Verbindungen erreicht ist. Dann müssen die Populationsgrößen einfach reduziert werden.